Расходные материалы и принадлежности на заказ по каталогам |
|
| Материалы и принадлежности Illumina на заказ | |
Разделы : |
|
| ПЦР цифровая | |
| Секвенаторы ДНК 2-го поколения NGS | |
| Электрофорез горизонтальный | |
| Электрофорез капиллярный на чипе | |
| Секвенирование NGS | |
| Эпигенетика | |
Товары |
|
|
001.24
|
|||||||||||
|
Набор для приготовления библиотек для секвенирования определённой (+ или -) цепи мРНК, включающий все необходимые компоненты.
Эти библиотеки предназначены для следующих задач:
В состав набора входят все необходимые компоненты для подготовки библиотеки:
Входящий в набор Poly(A) RNA Selection Kit (его можно также приобрести отдельно) обеспечивает эффективную элиминацию рибосомальной РНК. Доля ридов рРНК составляет не более 0.001%. |
|||||||||||
|
17005726
|
|||||||||||
|
Набор позволяет эффективно захватывать все подтипы РНК для составления единой библиотеки, даже при работе с образцами небольшой массы и невысокого качества:
Для получения оптимальных результатов мультиплексного секвенирования для работы с набором предназначены высококачественные уникальные парные праймеры с двойным индексированием (предлагаются в вариантах на 12 или 96 UDI-праймеров). |
|||||||||||
|
015.24
|
|||||||||||
Наборы QuantSeq 3’ mRNA-Seq предназначены для получения Illumina-совместимых библиотек из поли-А РНК. Протокол занимает около 4,5 часов. Особенность методики — получение только одного фрагмента с одного транскрипта, что даёт возможность оценить уровень экспрессии с высокой точностью. При этом секвенируются последовательности в непосредственной близости от 3’-конца мРНК. Наборы QuantSeq доступны как для транскриптов с прямой цепи (FWD), так и для обратной цепи (REV). Обе версии обеспечивают высокую специфичность по отношению к направлению цепи. В состав набора QuantSeq FWD входит праймер для синтеза второй цепи, содержащий линкерную последовательность Read 1. Эта последовательность обеспечивает генерацию ридов при секвенировании в направлении 3’-конца, т.е. соответствующих прямой последовательности мРНК. Более длинные риды дают более полную последовательность 3’-концов. Не рекомендуется секвенировать библиотеки QuantSeq FWD по методу парных прочтений (paired-end), так как линкерная последовательность Read 2 начинается с поли-Т, а гомополимеры снижают качество ридов. В состав набора входят магнитные частицы для очистки библиотек, что делает возможным автоматизацию процесса. Мультиплексирование до 96 образцов обеспечивается с помощью индексов i7, входящих в набор. Дополнительно можно использовать набор индекcов i5 (каталожный номер 047), в итоге можно объединить в библиотеке до 9216 образцов. В набор QuantSeq 3‘ mRNA-Seq Library Prep Kit (FWD) HT кроме i7, включены индексы i5 (5001-5004). В качестве стартового материала используется суммарная РНК, обогащение полиА-транскриптами происходит за счёт олиго-дТ праймеров. |
|||||||||||
|
016.24
|
|||||||||||
Наборы QuantSeq 3’ mRNA-Seq предназначены для получения Illumina-совместимых библиотек из поли-А РНК. Протокол занимает около 4,5 часов. Особенность методики — получение только одного фрагмента с одного транскрипта, что даёт возможность оценить уровень экспрессии с высокой точностью. При этом секвенируются последовательности в непосредственной близости от 3’-конца мРНК. Наборы QuantSeq доступны как для транскриптов с прямой цепи (FWD), так и для обратной цепи (REV). Обе версии обеспечивают высокую специфичность по отношению к направлению цепи. В состав набора QuantSeq REV входит олиго-дТ праймер, вводящий линкерную последовательность Read 1 в состав синтезируемой цепи. Этой последовательности комплементарен праймер CSP (Custom Sequencing Primer), входящий в набор и дающий последовательность, соответствующую кДНК. Библиотеки, полученные с помощью QuantSeq REV, можно секвенировать по методу парных прочтений (paired-end). В состав набора входят магнитные частицы для очистки библиотек, что делает возможным автоматизацию процесса. Мультиплексирование до 96 образцов обеспечивается с помощью индексов i7, входящих в набор. Дополнительно можно использовать набор индекcов i5 (каталожный номер 047), в итоге можно объединить в библиотеке до 9216 образцов. В качестве стартового материала используется суммарная РНК, обогащение полиА-транскриптами происходит за счёт олиго-дТ праймеров. |
|||||||||||
|
009.08
|
|||||||||||
|
Продукт доступен до 31.12.2019.
Набор для приготовления цепь-специфичных библиотек для секвенирования РНК, включающий все необходимые компоненты. Библиотеки предназначены для следующих задач:
С помощью набора SENSE можно получать библиотеки для секвенирования только на платформе Illumina. Исходным материалом для получения библиотек является РНК после удаления рибосомальной РНК или обогащения поли-А+. Для удаления рибосомальной РНК из образца можно использовать RiboCop rRNA Depletion Kit. Для работы с набором предлагаются два протокола - для интактной РНК и деградированной РНК (например, из FFPЕ-образцов). В состав набора входят все необходимые компоненты для подготовки библиотеки:
|
|||||||||||
|
052.08
|
|||||||||||
|
058.08
|
|||||||||||
|
080.96
|
|
96 реакций
|
По запросу По запросу |
|
|
||||||
|
081.96
|
|
96 реакций
|
По запросу По запросу |
|
|
||||||
|
020.96
|
|
96 реакций
|
По запросу По запросу |
|
|
||||||
|
024.96
|
|
96 образцов
|
По запросу По запросу |
|
|
||||||
|
026.96
|
|
96 образцов
|
По запросу По запросу |
|
|
||||||
|
028.96
|
|
96 образцов
|
По запросу По запросу |
|
|
||||||
|
033.24
|
|||||||||||
|
034.24
|
|||||||||||
|
035.24
|
|||||||||||
|
042.24
|
|||||||||||
|
ab185907-12
|
|||||||||||
|
ab185903-24
|
|
24 теста
|
хранение +4°C, транспортировка +4°C
|
По запросу По запросу |
|
|
|||||
|
ab185905
|
|
24 теста
|
По запросу По запросу |
|
|
||||||
|
ab185906-24
|
|
24 теста
|
По запросу По запросу |
|
|
||||||
|
1863040
|
|
|
хранение -20°C
|
424 347
424 347 AMD
|
|
|
|||||
|
|
|||||||||||
|
SY-410-1003
|
|
|
Регистрационное удостоверение на медицинское изделие
Росздравнадзора
|
По запросу По запросу |
|
|
|||||
|
Настольный интегрированный инструмент для NGS, включающий в одном приборе блоки для подготовки образцов (образования кластеров), секвенирования и обработки данных и интерфейс для управления. Принцип секвенирования основан на технологии Solexa, включающей обогащение методом bridge-ПЦР на проточном чипе, секвенирование методом синтеза (SBS) с использованием флуоресцентно-меченных нуклеотидов и детекцию света флуоресценции от кластеров ДНК.
Основные применения:
|
|||||||||||
|
12011928
|
|
96 реакций
|
783 133
783 133 AMD
|
|
|
||||||
|
Двойные индексированные праймеры SEQuoia представляют собой высококачественные, готовые к использованию пары праймеров с индексами i5 и i7, которые предназначены для использования с наборами для подготовки библиотек SEQuoia Complete Stranded RNA Library для создания библиотек для мультиплексного секвенирования с использованием платформ Illumina. Нуклеотидная последовательность каждого набора праймеров сбалансирована для достижения оптимальных результатов мультиплексного секвенирования. Набор содержит 12 уникальных адаптеров с двойным индексом в 12 отдельных флаконах с объемом, достаточным для восьми реакций в каждом флаконе. |
|||||||||||
|
12011930
|
|
96 реакций
|
783 133
783 133 AMD
|
|
|
||||||
|
Двойные индексированные праймеры SEQuoia представляют собой высококачественные, готовые к использованию пары праймеров с индексами i5 и i7, которые предназначены для использования с наборами для подготовки библиотек SEQuoia Complete Stranded RNA Library для создания библиотек для мультиплексного секвенирования с использованием платформ Illumina. Нуклеотидная последовательность каждого набора праймеров сбалансирована для достижения оптимальных результатов мультиплексного секвенирования. Планшет содержит 96 уникальных адаптеров с двойным индексом, расположенных вдоль колонок в 96-луночном планшете для ПЦР. |
|||||||||||
|
17006487
|
|
24 реакции
|
2 135 042
2 135 042 AMD
|
|
|
||||||
|
Автономное решение для деплеции рибосомальной РНК после подготовки библиотеки. Позволяет уменьшить число фрагментов при картировании РНК, сохраняя больше фрагментов в транскриптоме. Особенности:
|
|||||||||||
|
025.03
|
|
1 пробирка
|
По запросу По запросу |
|
|
||||||
|
Набор для приготовления цепь-специфичных библиотек для секвенирования РНК, включающий все необходимые компоненты. Библиотеки предназначены для следующих задач:
С помощью набора SENSE можно получать библиотеки для секвенирования только на платформе Illumina. Исходным материалом для получения библиотек является РНК после удаления рибосомальной РНК или обогащения поли-А+. Для удаления рибосомальной РНК из образца можно использовать RiboCop rRNA Depletion Kit. Для работы с набором предлагаются два протокола - для интактной РНК и деградированной РНК (например, из FFPЕ-образцов). В состав набора входят все необходимые компоненты для подготовки библиотеки:
|
|||||||||||
|
050.01
|
|||||||||||
|
Набор для приготовления цепь-специфичных библиотек для секвенирования РНК, включающий все необходимые компоненты. Библиотеки предназначены для следующих задач:
С помощью набора SENSE можно получать библиотеки для секвенирования только на платформе Illumina. Исходным материалом для получения библиотек является РНК после удаления рибосомальной РНК или обогащения поли-А+. Для удаления рибосомальной РНК из образца можно использовать RiboCop rRNA Depletion Kit. Для работы с набором предлагаются два протокола - для интактной РНК и деградированной РНК (например, из FFPЕ-образцов). В состав набора входят все необходимые компоненты для подготовки библиотеки:
|
|||||||||||
|
051.01
|
|||||||||||
|
Набор для приготовления цепь-специфичных библиотек для секвенирования РНК, включающий все необходимые компоненты. Библиотеки предназначены для следующих задач:
С помощью набора SENSE можно получать библиотеки для секвенирования только на платформе Illumina. Исходным материалом для получения библиотек является РНК после удаления рибосомальной РНК или обогащения поли-А+. Для удаления рибосомальной РНК из образца можно использовать RiboCop rRNA Depletion Kit. Для работы с набором предлагаются два протокола - для интактной РНК и деградированной РНК (например, из FFPЕ-образцов). В состав набора входят все необходимые компоненты для подготовки библиотеки:
|
|||||||||||
|
006.08
|
|||||||||||
|
Набор для приготовления библиотек для секвенирования определённой (+ или -) цепи мРНК, включающий все необходимые компоненты.
Эти библиотеки предназначены для следующих задач:
В состав набора входят все необходимые компоненты для подготовки библиотеки:
Входящий в набор Poly(A) RNA Selection Kit (его можно также приобрести отдельно) обеспечивает эффективную элиминацию рибосомальной РНК. Доля ридов рРНК составляет не более 0.001%. |
|||||||||||
|
BLU0001
|
|
|
По запросу По запросу |
|
|
||||||
|
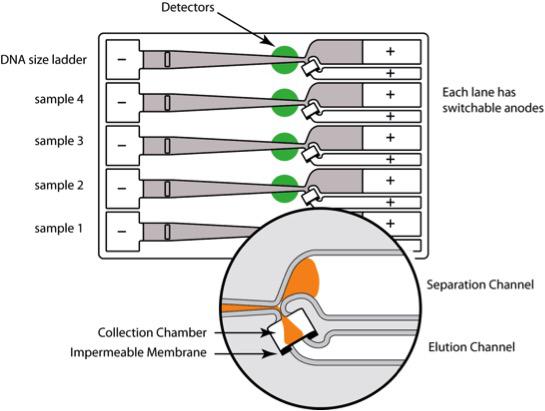
BluePippin — система препаративного электрофореза и пульс-электрофореза, позволяет разделять и выделять фрагменты НК, размером 90-50000 п.н., а так же белки массой 18-200 кДа.
Области применения:
Принцип работы: в системе используются готовые картриджи на пять изолированных капилляров, заполненных агарозным гелем разной процентной концентрации, которая выбирается пользователем в зависимости от величины фрагмента образца. В конце каждого капилляра имеется Y-образное разветвление. Оба выходящих капилляра имеют независимое подключение к катодам. Движение фрагментов ДНК в капилляре как и в «классической» методике происходит под действием тока, но при приближении фрагментов ДНК требуемого размера к месту разветвления, происходит переключение катодов, и искомый фрагмент попадает в боковое ответвление капилляра и собирается в канале для сборки. Для проведения электрофореза нуклеиновых кислот используется Tris-TAPS (или Трис-ТАПС) буфер, для белков — SDS-буфер. Программное обеспечение определяет время элюирования на основе миграции ДНК-маркера.
4 режима программирования:
3 стратегии детектирования фракций:
Преимущества метода:
Схема работы:
Информация о свежих статьях:
1. Nature Methods. Assembly and diploid architecture of an individual human genome via single-molecule technologies (июнь, 2015); Authors: Matthew Pendleton, Robert Sebra, Andy Wing Chun Pang, Ajay Ummat et al.
Видео: Для проведения пульс-электрофореза применяется специальный источник питания Pippin Pulse.
|
|||||||||||
|
|
|||||||||||
Все поля со звездочкой (*) обязательны для заполнения
Данное действие необратимо.
чтобы сделать работу на сайте еще удобнее!
С помощью личного кабинета Вы сможете:
- моментально получать счета на оформленные заказы;
- отслеживать статусы выполнения заказа по оплате, отгрузке, наличию товаров на складе;
- вести историю заказов, повторять заказы полностью или частично;
- выбирать персонального менеджера;
- формировать списки избранного среди товаров, справочных материалов и видео;
- делать заказ со страницы избранных товаров;
- экономить время при заполнении форм заказа по каталогам и регистрации на мероприятия.