|
Oxford Nanopore Technologies
|
|||||||||||||||||||||||||||||||
|
SQK-RBK004
|
|
|
хранение -20°C
|
По запросу По запросу |
|
|
|||||||||||||||||||||||||
|
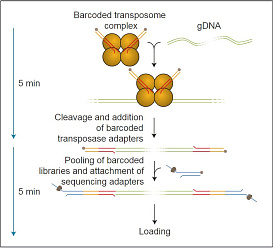
Набор предназначен для быстрого и простого получения сиквенсной библиотеки, включающей до 12 различных образцов ДНК. С помощью набора Rapid Barcoding Kit можно:
Объединение нескольких образцов, или мультиплексирование, возможно благодаря бар-кодированию разных ДНК. Это удобно в тех случаях, когда требуемый выход данных при секвенировании одного образца намного меньше максимального выхода, получаемого с одной проточной ячейки. В состав набора входит 12 бар-кодов, таким образом, в одну библиотеку можно загрузить до 12 образцов ДНК. Это позволяет снизить стоимость секвенирования одного образца почти в 10 раз.
Особенности набора:
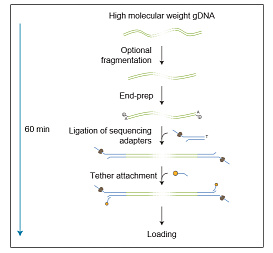
* Полные протоколы доступны на сайте www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community. Набор предназначен для исследований, требующих простой пробоподготовки и быстрого результата, но не максимальной производительности секвенирования. Так как количество стадий пипетирования и время подготовки минимальны, набор даёт возможность получения длинных ридов, но макcимальная длина зависит от качества исходной ДНК. Поэтому для лучшего результата рекомендуется использовать ДНК со средней длиной фрагментов не менее 30 гигабаз. Для выделения высокомолекулярной геномной ДНК можно использовать набор Blood & Cell Culture DNA Mini Kit (Qiagen). Пробоподготовка заключается в расщеплении ДНК транспозазой и присоединении к фрагментам бар-кодов. Баркодированные фрагменты ДНК объединяются, и к ним присоединяются сиквенсные адаптеры. Рекомендуемое исходное количество ДНК — не менее 400 нг. Возможно получение библиотеки из меньшего количества ДНК, а также из фрагментированной ДНК, но производительность будет ниже. Для получения библиотеки из меньших количеств ДНК рекомендуется набор PCR Sequencing Kit и протоколы Low Input и Rapid Low Input*. Так как при получении библиотеки отсутствует стадия ПЦР, данный набор пригоден для выявления модифицированных нуклеотидов. Сортировка ридов в соответствии с баркодами производится с помощью программных инструментов, доступных в рамках платформы EPI2ME. В состав Rapid Barcoding Kit также входит набор Flow Cell Priming Kit для загрузки библиотеки в поточную ячейку. Сиквенсные адаптеры RAP, входящие в набор, также могут быть приобретены в виде отдельного набора EXP-RAP001.
Условия хранения: -20 °С.
Дополнительные расходные материалы и реактивы:
Опционально:
Дополнительное оборудование:
Опционально:
|
|||||||||||||||||||||||||||||||
|
SQK-RPB004
|
|
|
хранение -20°C, транспортировка +2...+8 °C
|
По запросу По запросу |
|
|
|||||||||||||||||||||||||
|
Набор предназначен для быстрого и простого получения сиквенсной библиотеки, включающей до 12 различных образцов ДНК, в тех случаях, когда количество ДНК
ограничено. С помощью набора Rapid PCR Barcoding Kit можно:
Особенности набора:
* Полные протоколы доступны на сайте www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community. Набор предназначен для исследований, требующих простой пробоподготовки и быстрого результата, но не максимальной производительности секвенирования. Пробоподготовка заключается в расщеплении ДНК транспозазой и присоединении к фрагментам олигонуклеотидов, содержащих сайты связывания ПЦР-праймеров. Затем каждый образец ДНК амплифицируется с помощью баркодированных праймеров. Баркодированные ампликоны объединяются, и к ним присоединяются сиквенсные адаптеры. Длина ампликонов в среднем составляет около 2 килобаз и не зависит от длины исходной ДНК. Это связано в большей степени с особенностями пробоподготовки, а не с процессивностью Taq-полимеразы. Поэтому для пользователей, работающих с малым исходным количеством геномной ДНК и желающих получить риды максимальной длины, рекомендуется набор Ligation Sequencing Kit и протокол Low input genomic DNA with PCR. Набор позволяет работать с ДНК, содержащей примеси, которые снижают эффективность получения библиотеки. Это происходит благодаря стадии ПЦР. Но необходимо учитывать, что амплификация разных фрагментов может идти с разной эффективностью. Если исходное количество ДНК менее 1 пг, рекомендуется полногеномная амплификация. Сортировка ридов в соответствии с баркодами производится с помощью программных инструментов, доступных в рамках платформы EPI2ME. В состав Rapid PCR Barcoding Kit также входит набор Flow Cell Priming Kit для загрузки библиотеки в поточную ячейку. Условия хранения: -20 °С. Дополнительные расходные материалы и реактивы:
Дополнительное оборудование:
|
|||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
|
|||||||||||||||||||||||||||||||
|
SQK-RAD004
|
|
|
хранение -20°C
|
По запросу По запросу |
|
|
|||||||||||||||||||||||||
|
Набор предназначен для быстрого получения библиотеки из геномной ДНК с помощью простого 2-шагового протокола, пробоподготовка занимает не более 10 минут. Процесс основан на транспозазном расщеплении ДНК с последующей пришивкой сиквенсных адаптеров.
Особенности набора:
Набор предназначен для исследований, требующих простой пробоподготовки и быстрого результата, но не максимальной производительности секвенирования. Так как количество стадий пипетирования и время подготовки минимальны, набор даёт возможность получения длинных ридов, но макcимальная длина зависит от качества исходной ДНК. Поэтому для лучшего результата рекомендуется использовать ДНК со средней длиной фрагментов не менее 30 гигабаз. Для выделения высокомолекулярной геномной ДНК можно использовать набор Blood & Cell Culture DNA Mini Kit (Qiagen). Рекомендуемое исходное количество ДНК — не менее 400 нг. Возможно получение библиотеки из меньшего количества ДНК, а также из фрагментированной ДНК, но производительность будет ниже. Для получения библиотеки из меньших количеств ДНК рекомендуется набор PCR Sequencing Kit и протоколы Low Input и Rapid Low Input. Так как при получении библиотеки отсутствует стадия ПЦР, данный набор пригоден для выявления модифицированных нуклеотидов. Дополнительные расходные материалы:
Дополнительное оборудование:
Опционально:
|
|||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
|
|||||||||||||||||||||||||||||||
|
SQK-PBK004
|
|
|
хранение -20°C
|
По запросу По запросу |
|
|
|||||||||||||||||||||||||
|
Мультиплексное секвенирование с баркодированием удобно, если количество сиквенсных данных, получаемое с одного образца, существенно меньше производительности проточной ячейки. В таком случае имеет смысл объединять образцы в одну библиотеку и секвенировать их одновременно. Это также снижает стоимость секвенирования каждого образца.
Набор PCR Barcoding Kit удобен в тех случаях, когда:
Особенности набора:
* Полные протоколы доступны на сайте
www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community. Пробоподготовка заключается в опциональной фрагментации геномной ДНК с последующей достройкой концов, лигировании адаптеров к достроенным концам и ПЦР с праймерами, содержащими бар-коды. В состав набора входит 12 пар бар-кодированных праймеров. В конечном итоге образцы после ПЦР объединяются, и к смеси добавляются сиквенсные адаптеры. Длины фрагментов, получаемых в ходе ПЦР, зависят от длины исходной матрицы и от процессивности ДНК-полимеразы. Максимальная длина рида составляет в среднем около 2 тыс п.н. Пользователь также может использовать метод ПЦР с 2 парами праймеров (nested PCR); при этом одна пара праймеров - пользовательские праймеры к определённой последовательности ДНК, а вторая - бар-кодированные праймеры из набора. 5’-концы пользовательских праймеров должны быть комплементарны 3’-концам бар-кодированных праймеров. Последовательности бар-кодов автоматически распознаются программой для интерпретации данных секвенирования, и риды группируются в соответствии с бар-кодами. Ещё одно преимущество набора — если исходная ДНК загрязнена, то в ходе ПЦР существенно снижается концентрация примесей, которые могут ингибировать процесс получения библиотеки. Если количество исходной ДНК менее 1 нг — рекомендуется предварительная полногеномная амплификация. Дополнительные расходные материалы и реактивы:
Опционально:
Дополнительное оборудование:
|
|||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
|
|||||||||||||||||||||||||||||||
|
SQK-DCS109
|
|
|
хранение -20°C
|
Регистрационное удостоверение на медицинское изделие
Росздравнадзора
|
По запросу По запросу |
|
|
||||||||||||||||||||||||
|
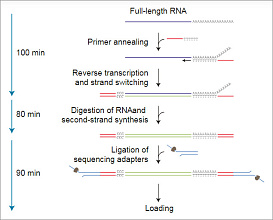
Direct cDNA Sequencing Kit — набор для прямого секвенирования кДНК. Набор предназначен для секвенирования РНК через стадию синтеза кДНК без стадии ПЦР. С помощью набора Direct DNA Sequencing Kit можно:
Особенности набора:
Набор рассчитан на работу с поли-А+ РНК, но возможно также использование РНК со специфическими 3’-концевыми последовательностями (например, вирусной РНК). Для этого необходимы олигонуклеотиды, комплементарные этим последовательностям. Пробоподготовка заключается в синтезе комплементарной цепи кДНК с помощью обратной транскрипции. Обогащение библиотеки полноразмерными копиями происходит за счёт переключения цепей при обратной транскрипции (strand-switching). Затем цепь РНК расщепляется, и достраивается вторая цепь кДНК. Сиквенсные адаптеры лигируются на двухцепочечную кДНК (в нанопоры может проходить любая из цепей кДНК). При работе с Direct cDNA Sequencing Kit предусмотрена возможность мультиплексирования с помощью наборов Native Barcoding Expansion 1-12 (EXP-NBD104) или Native Barcoding Expansion 13-24 (EXP-NBD105). В одну библиотеку может быть загружено до 12 образцов кДНК. Дополнительные расходные материалы и реактивы:
Дополнительное оборудование:
Опционально: |
|||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
|
|||||||||||||||||||||||||||||||
|
SQK-RNA002
|
|
|
хранение -20°C, транспортировка +2...+8 °C
|
По запросу По запросу |
|
|
|||||||||||||||||||||||||
|
Набор предназначен для прямого секвенирования РНК без стадии обратной транскрипции. С помощью набора Direct RNA Sequencing Kit можно:
Особенности набора
* Полные протоколы доступны на сайте www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community.
Набор предназначен для работы с молекулами РНК, обладающими 3’-концевыми полиА –структурами — эукариотическими мРНК, полиаденилированными вирусными РНК или РНК, обработанными наборами для полиаденилирования. Для РНК без полиА-концов можно синтезировать пользовательские адаптеры, комплементарные 3’-концам (например, для тРНК или 16S-РНК). В отличие от протоколов секвенирования кДНК, в случае использования набора Direct RNA Sequencing Kit происходит секвенирование нативных молекул РНК; только они проходят через поры проточной ячейки. Стадия обратной транскрипции при пробоподготовке опциональна, но настоятельно рекомендуется — это повышает производительность секвенирования. Набор Direct RNA Sequencing Kit незаменим в случае анализа модифицированных нуклеотидов. Если задача выявления таких нуклеотидов не ставится, рекомендуются наборы для секвенирования кДНК, так как они обеспечивают более высокую производительность.
Дополнительные расходные материалы и реактивы:
Дополнительное оборудование:
Опционально:
|
|||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
|
|||||||||||||||||||||||||||||||
|
SQK-RAB204
|
|
|
хранение -20°C
|
По запросу По запросу |
|
|
|||||||||||||||||||||||||
|
Набор предназначен для секвенирования бактериальных генов, кодирующих рибосомальную РНК 16S, являющуюся видоспецифичной. С помощью набора 16S Barcoding Kit можно:
Объединение нескольких образцов, или мультиплексирование, возможно благодаря баркодированию разных ДНК. Это удобно в тех случаях, когда требуемый выход данных при секвенировании одного образца намного меньше максимального выхода, получаемого с одной проточной ячейки. В состав набора входит 12 бар-кодов, таким образом, в одну библиотеку можно загрузить до 12 образцов ДНК. Это позволяет снизить стоимость секвенирования одного образца почти в 10 раз. Особенности набора:
* Полные протоколы доступны на сайте www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community. Пробоподготовка заключается в амплификации гена 16S со специфических праймеров (27F и 1492R), содержащих бар-коды. Затем к 5’-концам ампликонов лигируются сиквенсные адаптеры. Анализ результатов секвенирования библиотеки, полученной с помощью 16S Barcoding Kit, проводится на платформе EPI2ME с помощью специального ПО, сортирующего риды в соответствии с баркодированием и идентифицирующего их видовую принадлежность. В состав 16S Barcoding Kit также входит набор Flow Cell Priming Kit для загрузки библиотеки в поточную ячейку. Условия хранения: - 20 °С. Дополнительные расходные материалы и реактивы:
Дополнительное оборудование:
Опционально:
|
|||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
|
|||||||||||||||||||||||||||||||
|
SQK-LRK001
|
|
|
По запросу По запросу |
|
|
||||||||||||||||||||||||||
|
Набор предназначен для получения библиотеки из геномной ДНК в полевых условиях, когда ограничена возможность хранения реагентов при низких температурах. Процесс пробоподготовки основан на транспозазном расщеплении ДНК с последующей пришивкой сиквенсных адаптеров и занимает не более 10 минут.
Особенности набора:
Набор оптимизирован для хранения при температуре окружающей среды, так как реагенты в его составе находятся в лиофилизированном виде. Количество стадий пипетирования и время подготовки минимальны, поэтому набор даёт возможность получения длинных ридов, но максимальная длина зависит от качества исходной ДНК. Рекомендуемое исходное количество ДНК — не менее 400 нг. Возможно получение библиотеки из меньшего количества ДНК, а также из фрагментированной ДНК, но производительность будет ниже. Так как при получении библиотеки отсутствует стадия ПЦР, данный набор пригоден для выявления модифицированных нуклеотидов. В состав Field Sequencing Kit также входит набор Flow Cell Priming Kit для загрузки библиотеки в поточную ячейку. Дополнительные расходные материалы:
|
|||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
|
|||||||||||||||||||||||||||||||
|
SQK-LSK109
|
|
|
хранение -20°C, транспортировка +2...+8 °C
|
По запросу По запросу |
|
|
|||||||||||||||||||||||||
|
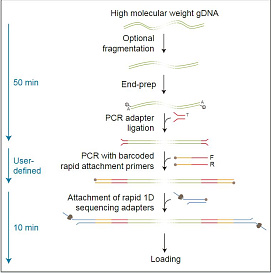
Набор предназначен для секвенирования двухцепочечной ДНК. Это наиболее универсальный набор из всех, предлагаемых Oxford Nanopore Technologies. С помощью набора Ligation Sequencing Kit можно:
Для данного набора предлагается множество протоколов пробоподготовки библиотек из различного исходного материала (геномная ДНК, плазмидная ДНК, ампликоны) и для различных задач (геномное секвенирование de novo, ресеквенирование, таргетное секвенирование, мультиплексное секвенирование и т.д.). Процесс пробоподготовки заключается в достройке концов ДНК, пришивке 3’-концевой dA и лигировании к ней адаптеров. Особенности набора
* Полные протоколы доступны на сайте www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community. Набор оптимизирован для высокопроизводительного секвенирования. Для максимальной производительности рекомендуется получать библиотеку из 100-200 фмоль высокомолекулярной ДНК. Также важна степень чистоты ДНК, загрязнения существенно снижают производительность. Рекомендуется использовать наноспектрофотометр для оценки степени чистоты ДНК по следующим критериям:
Можно секвенировать библиотеки, полученные с помощью Ligation Sequencing Kit из меньшего количества ДНК или ДНК низкого качества, но это существенно уменьшает производительность секвенирования. Частично можно улучшить библиотеку с помощью стадии ПЦР: происходит обогащение библиотеки при малом стартовом количестве ДНК, а также снижается влияние примесей за счёт разведения образца. Но, если ПЦР является неотъемлемой частью пробоподготовки, рекомендуется использовать PCR Sequencing Kit или Rapid PCR Barcoding Kit. Аналогично, если планируется регулярно секвенировать ампликоны или кДНК, рекомендуется использовать соответствующие наборы — PCR Sequencing Kit или, для секвенирования кДНК, cDNA-PCR Sequencing Kit и Direct cDNA Sequencing Kit. Набор Ligation Sequencing Kit может быть использован для мультиплексного секвенирования. Для этого необходимы дополнительные наборы Native Barcoding Expansion 1-12 и 13-24 (для мультиплексирования без стадии ПЦР) или PCR Barcoding Expansion 1-12 и 1-96 (для мультиплексирования со стадией ПЦР). Дополнительные расходные материалы и реактивы:
Опционально:
Дополнительное оборудование:
Опционально:
|
|||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
|
|||||||||||||||||||||||||||||||
|
SQK-PSK004
|
|
|
хранение -20°C, транспортировка +2...+8 °C
|
По запросу По запросу |
|
|
|||||||||||||||||||||||||
|
Набор предназначен для получения сиквенсной библиотеки из геномной ДНК в тех случаях, когда количество стартового материала ограничено. С помощью набора PCR Sequencing Kit можно:
Особенности набора:
* Полные протоколы доступны на сайте www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community. Пробоподготовка заключается в опциональной фрагментации ДНК с последующей достройкой концов до тупых и присоединением 3’-dA. К 3’-dA лигируются олигонуклеотиды, содержащие сайты связывания ПЦР-праймеров. ДНК амплифицируется, и к ампликонам присоединяются сиквенсные адаптеры. Стадия ПЦР зависит от исходного количества ДНК и желаемой длины ампликонов. Рекомендуемый протокол ПЦР рассчитан на 100 нг исходной ДНК. Длина рида составляет в среднем около 2 килобаз и зависит не столько от процессивности Taq-полимеразы, сколько от особенностей пробоподготовки. Но при этом максимальная длина рида выше, чем при использовании набора Rapid PCR Barcoding Kit и может достигать 10 килобаз. Пользователь также может использовать метод ПЦР с 2 парами праймеров (nested PCR); при этом одна пара праймеров — пользовательские праймеры к определённой последовательности ДНК, а вторая — бар-кодированные праймеры из набора. 5’-концы пользовательских праймеров должны быть комплементарны 3’-концам бар-кодированных праймеров. Набор позволяет работать с ДНК, содержащей примеси, которые снижают эффективность получения библиотеки. Это происходит благодаря стадии ПЦР. Но необходимо учитывать, что амплификация разных фрагментов может идти с разной эффективностью. Если исходное количество ДНК менее 1 пг, рекомендуется полногеномная амплификация. В состав PCR Sequencing Kit также входит набор Flow Cell Priming Kit для загрузки библиотеки в поточную ячейку. Условия хранения: -20 °С. Дополнительные расходные материалы и реактивы:
Опционально:
Дополнительное оборудование:
Опционально:
|
|||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
|
|||||||||||||||||||||||||||||||
|
SQK-PCS109
|
|
|
хранение -20°C
|
По запросу По запросу |
|
|
|||||||||||||||||||||||||
|
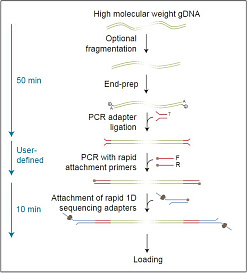
Набор для секвенирования кДНК со стадией ПЦР PCR-cDNA Sequencing Kit предназначен для секвенирования РНК через стадию синтеза кДНК с последующей ПЦР. С помощью набора PCR-cDNA Sequencing Kit можно:
В состав набора входит Pro Flow Cell Priming Kit для загрузки библиотек в ячейку, совместимый с ячейками для PromethION Особенности набора:
Набор рассчитан на работу с минимальным количеством поли-А+ РНК, для получения библиотеки достаточно всего 1 нг. В случае невозможности выделения полиА+-фракции допустимо использование суммарной РНК (рекомендуется не менее 50 нг), но в этом случае требуется оптимизация пробоподготовки. Пробоподготовка заключается в синтезе комплементарной цепи кДНК с помощью обратной транскрипции. Обогащение библиотеки полноразмерными копиями происходит за счёт переключения цепей при обратной транскрипции (strand-switching). Затем происходит синтез двухцепочечной кДНК с помощью ПЦР, в реакции используются специфические праймеры для последующего безлигазного присоединения сиквенсных адаптеров. При работе с PCR-cDNA Sequencing Kit предусмотрена возможность мультиплексирования с помощью набора PCR Barcoding Expansion (SQK-PBK004). В одну библиотеку может быть загружено до 12 образцов кДНК. Дополнительные расходные материалы и реактивы:
Дополнительное оборудование:
Опционально: |
|||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||
Все поля со звездочкой (*) обязательны для заполнения
Данное действие необратимо.
чтобы сделать работу на сайте еще удобнее!
С помощью личного кабинета Вы сможете:
- моментально получать счета на оформленные заказы;
- отслеживать статусы выполнения заказа по оплате, отгрузке, наличию товаров на складе;
- вести историю заказов, повторять заказы полностью или частично;
- выбирать персонального менеджера;
- формировать списки избранного среди товаров, справочных материалов и видео;
- делать заказ со страницы избранных товаров;
- экономить время при заполнении форм заказа по каталогам и регистрации на мероприятия.